0
留言稍后联系!

发布时间:2022-04-12所属分类:农业论文浏览:1602次
摘 要: 摘 要 :目前 GenBank 数据库共收录 167 种直翅目昆虫全线粒体基因组序列,涉及蝗亚目 9 个总科 22 个科 99 个物种,螽亚目 7 个总科 12 个科 68 个物种。在此基础上,该文分析了直翅目昆虫线粒体基因组的基本特征,概述了线粒体全基因组在直翅目昆虫系统发育研究上的

摘 要 :目前 GenBank 数据库共收录 167 种直翅目昆虫全线粒体基因组序列,涉及蝗亚目 9 个总科 22 个科 99 个物种,螽亚目 7 个总科 12 个科 68 个物种。在此基础上,该文分析了直翅目昆虫线粒体基因组的基本特征,概述了线粒体全基因组在直翅目昆虫系统发育研究上的应用 ;同时基于线粒体全基因组序列重建了直翅目昆虫的系统发育关系。主要结果如下 :(1) 直翅目昆虫存在 8 种线粒体基因组排列类型,其中 trnK-trnD 重排现象仅发生在蝗总科中,trnN-trnS-trnE 重排现象仅发生在蟋蟀总科中,trnM-trnI-(-trnQ) 重排现象仅发生在拟叶蟲亚科中 ;(2) 直翅目昆虫全线粒体基因组的碱基组成具有明显的 AT 偏向性 ;(3) 不同的蛋白质编码基因在直翅目昆虫中的进化速率不同 ;(4) 支持直翅目以及螽亚目和蝗亚目的单系性 ;(5) 不支持沙螽总科单系性;(6) 支持蝗亚目各总科阶元的单系性,且各总科间的系统发育关系为:( 蚤蝼总科 + ( 蚱总科 + ( 䗛蜢总科 + ( 蜢总科 + ( 长角蝗总科 + ( 牛蝗总科 + 叶翅蝗总科 ) + ( 锥头蝗总科 + 蝗总科 ))))))。
关键词 :直翅目 ;线粒体基因组 ;比较基因组 ;进化
直翅目 (Orthoptera) 隶属于节肢动物门 (Arthropoda) 昆虫纲 (Insecta),目前全世界已描述的种类有 27 941 种 (http://orthoptera.speciesfile.org/HomePage/ Orthoptera/HomePage.aspx)。直翅目昆虫几乎占据着整个地球上除极地以外的所有陆生栖息地,在生态系统中均发挥着重要的作用,有很多物种被作为解剖学、生态学、生物化学、生物声学以及进化生物学等领域的模式生物 [1-6]。目前,国际上普遍认同将直翅目分为蝗亚目(Caelifera)和螽亚目(Ensifera),但有关亚目下高级阶元的划分仍然存在分歧 [7]。准确鉴定直翅目昆虫种类,深入了解直翅目昆虫的起源进化、系统发育以及种群扩张,对直翅目害虫防控、经济种质资源利用具有重要的指导意义。
昆虫线粒体基因组为环状双链的共价闭合 DNA 分子,具有相对分子质量小、结构简单、高拷贝、基因排列相对保守、基因进化速率快以及物种内几乎不发生重组的母系遗传特点,目前广泛应用于昆虫物种鉴定、分子系统发生学、生物地理学以及种群遗传结构等领域的研究 [8-11]。近年来,随着高通量测序的发展,科研人员获得了越来越多的直翅目昆虫线粒体基因组序列,为从基因组水平上研究直翅目昆虫进化提供了良好的契机。
本文汇总了过去 22 年间直翅目昆虫线粒体基因组测序的成果以及相关文献,总结并比较了基因组大小、碱基组成、密码子使用、基因进化速率、基因重排及特殊结构、RNA 基因及控制区的基本特征,并分析构建了直翅目系统发育树,为系统研究直翅目昆虫的分类及系统发生积累了基础资料。
1 直翅目昆虫线粒体基因组测序现状
1995 年,Flook 等 [12] 发表了第一个直翅目昆虫非洲飞蝗 (Locusta migratoria) 的线粒体基因组全序列。随着测序技术的不断发展,截至 2017 年 10 月 13 日,GenBank 数据库共收录 167 种直翅目昆虫全线粒体基因组序列。其中来自蝗亚目的物种最多,共计 99 种,涉及 9 个总科 22 个科 ;来自螽亚目的物种共计 68 种,涉及 7 个总科 12 个科。其中本实验室共提交 53 种 (31.74%) 直翅目昆虫线粒体基因组,为重建直翅目昆虫类群间系统发育关系,揭示直翅目昆虫的进化过程提供了丰富的基础分子资源 ( 附表 1)。虽然目前已获得全长线粒体基因组的物种已覆盖直翅目昆虫的所有总科,但与庞大的直翅目昆虫家族相比,测序的物种还微乎其微,例如怪螽科 Cooloolidae、癞蟋科 Mogoplistidae 和莫蜢科 Morabidae 等部分科级阶元尚无代表物种。
2 直翅目昆虫线粒体基因组基本特征
2.1 线粒体基因组大小
与大多数后生动物线粒体基因组相似,目前已测得的所有直翅目昆虫全线粒体基因组均为闭合环状双链 DNA,并编码 13 个蛋白质编码基因、2 个 rRNA 基因和 22 个 tRNA 基因。其中粒体基因组最小的为 Hemicharilaus monomorphus[13],长度为 13 291 bp ;最大的线为纤细绿露螽 (Phaneroptera gracilis),长度为 18 255 bp。直翅目昆虫线粒体基因组大小的变化,主要受控制区或基因组间隔区的数量和长度影响,例如疑钩顶螽 (Ruspolia dubia) [14] 的控制区长度仅有 70 bp ;而基因组长度最长的纤细绿露螽除了具有一个长度为 1 548 bp 的控制区,在 nad2 和 trnW 之间还具有一个长度为 1 830 bp 的基因间隔区。
2.2 基因重排与特殊结构
根据基因位置和转录方向的变化,昆虫线粒体基因重排可分为易位 ( 基因位置改变,转录方向不变 )、倒位 ( 基因位置不变,转录方向改变 ) 和基因洗牌 ( 多个基因发生易位和 ( 或 ) 倒位 ) 三种主要的类型 [15]。目前已测线粒体基因组的直翅目昆虫存在 8 种线粒体基因组排列类型 :有 54 种直翅目昆虫以图 1-1 所示的典型的节肢动物门线粒体基因组方式排列,包括大多数螽亚目昆虫 (50 种 ) 和极少量的蝗亚目昆虫 (4 种 ) ;有 90 种直翅目昆虫线粒体基因组中 trnK 与 trnD 基因发生易位,并以图 1-2 所示的 trnD-trnK 方式排列 ( 也称 KD 重排 ),该现象广泛存在于除蚤蝼总科和蜢总科的变色乌蜢 (Erianthus versicolor) [13] 以外的蝗亚目昆虫中,而螽亚目昆虫均无 KD 重排现象;有 9 种直翅目昆虫线粒体基因组中 trnN、trnS 与 trnE 基因发生基因洗牌,并以图 1-3 所示的 (-trnE)-(-trnS)-(-trnN)、(-trnE)- (trnS)-(-trnN) 或 (-trnE)-( trnS)-(trnN) 三种方式存在,该现象存在于所有的蟋蟀总科中 ;有 3 种直翅目昆虫线粒体基因组中 trnI、trnQ 与 trnM 基因发生基因洗牌,并以图 1-4 所示的 trnM-trnI-(-trnQ) 方式存在,该现象仅存在于拟叶蟲亚科 (Pseudophyllinae) 中 ;有 3 种直翅目昆虫线粒体基因组中 AT 富集区、trnI、trnQ、trnM 与 nd2 基因发生基因洗牌,并以图 1-5 所示的 trnI-trnM-nd2-AT-(-trnQ) 方式存在,该现象仅存在于露螽科的傅氏绿露螽 (Holochlora fruhstorferi)、长裂华绿露螽 (Sinochlora longifissa) [16]、四川华绿露螽 (Sinochlora szechwanensis) 中 ;鸣螽科的 Cyphoderris monstrosa[6] 线粒体基因组中 trnA 和 trnR 基因发生易位,并以图 1-6 所示的 trnR-trnA 方式存在 ;螽斯科的三锥迟螽 (Lipotactes tripyrga) 线粒体基因组中 trnG、nd3、trnA、trnR、trnN 和 trnS 基因发生基因洗牌,并以图 1-7 所示的 trnRtrnS-trnA-trnN-trnG-nd3 的方式存在 ;露螽科的污翅糙颈露螽 (Ruidocollaris obscura) [17] 线粒体基因组中 AT 富集区通过基因洗牌出现在 trnY 和 cox1 之间,并以图 1-8 所示的 (-trnY)-AT-cox1 的方式存在。
此外有 4 种已测直翅目昆虫中存在特殊的线粒体基因组结构,分别为锥头蝗科的金澜沧蝗 (Mekongiella kingdoni) [18] 缺少 trnR 基因,裂趾螽科的 Comicus campestris[6] 缺少 trnM 基因,剑角蝗科的 Locusta migratoria migratoria 和驼峰科的 Troglophilus neglectus[19] 在 cox1 和 cox2 基因存在 2 个 trnL。总体来说,相对于膜翅目等复新翅目昆虫 [20],直翅目昆虫线粒体基因组重排事件相对保守,除了蝗亚目的 KD 重排,仅有 23 种直翅目昆虫线粒体基因组存在特殊结构或发生了重排。目前,对基因重排现象的解释主要有复制非随机丢失模型、复制随机删除模型、重组以及由 tRNA 基因错误起始引起的复制 4 种解释 [21]。在这些模型中,复制随机删除模型已经获得部分证据的支持,但至今仍然没有一种完美的模型可以解释所有的重排现象 [22]。因此,对于高度重排的线粒体基因组可以用多个模型解释,例如,对于仅发生 KD 重排现象的蝗亚目可以用复制随机删除模型解释,而对于发生了基因洗牌的其他类型的线粒体重排现象就需要结合复制删除模型及重组模型来共同解释。
2.3 碱基组成与密码子使用情况
已测的直翅目昆虫全线粒体基因组具有明显的 AT 偏向性 ( 附表 2),平均 A+T 含量为 73.31%,其中 A+T 含量最高的为蜢科的 Paramastax nigra[13] (78.00%),最低为优雅蝈螽 (Gampsocleis gratiosa) [23] (65.30%)。
对直翅目亚目及总科阶元的全线粒体基因组的平均 AT 含量分析比较发现 :2 个亚目中,蝗亚目的 A+T 含量 (74.24%) 高于螽亚目 (71.95%) ;蝗亚目的 9 个总科中,叶翅蝗总科的 A+T 含量最高 (78.00%),而蚤蝼总科最低 (68.80%) ;螽亚目的 7 个总科中,裂跗螽总科 A+T 含量最高 (75.00%),而原螽总科最低 (69.45%)。一般来说,同一科级阶元中的物种间碱基含量变异较小,例如在蝗亚目中, A+T 含量在测序物种量最多的剑角蝗科 (SE=0.12) 和癞蝗科 (SE=0.15) 中呈现出明显的稳态 ;但并非总是如此,例如在螽亚目中,测序物种量最多的螽斯科 (SE=0.48)、露螽科 (SE=0.33) 和蟋蟀科 Gryllidae (SE=0.63) 中,不同的物种间 AT 含量差异较大 ( 图 2),类似的现象也出现在半翅目中 [22]。
除了日本纺织娘 (Mecopoda niponensis) [24]、傅氏绿露螽 (Holochlora fruhstorferi) [11]、四川华绿露螽 (Sinochlora szechwanensis)、黑角绿露螽 (Phaneroptera nigroantennata)、Tridactylus sp. 和 Mirhipipteryx andensis[6] 外,所有已测直翅目昆虫线粒体全基因组的 4 种碱基含量均为 As>Ts>Cs>Gs,同时还呈现出较强的 A 碱基和 C 碱基偏好 (AT-Skew = 0.11, GC-Skew = -0.21)。直翅目昆虫线粒体基因组呈现出的碱基组成偏好性与在进化的过程中突变、自然选择、随机遗传漂变、水平基因转移和基因组结构等多因素相互作用相关,导致线粒体基因组碱基含量A>T和C>G,碱基组成偏向于A和C两种碱基[25],符合一般后生动物线粒体基因组 AT 正偏差和 GC 负偏差的特点 [26]。
对直翅目昆虫亚目及总科阶元的全线粒体基因组碱基组成的分析比较发现 ( 图 3) :蝗亚目和螽亚目的碱基偏斜均符合后生动物线粒体基因组的普遍规律 ;蝗亚目的碱基组成偏斜最强烈 (AT skew = 0.15, GC skew = -0.18),螽亚目正链的 AT 碱基组成最为接近 (AT skew = 0.05, GC skew = -0.27)。蝗亚目 9 个总科的碱基偏斜均符合普遍规律,除蚤蝼总科正链的AT碱基组成最为接近(AT skew = 0.02)外,其余 8 个总科 AT 偏斜相对均很强烈,AT 偏斜范围为0.11~0.23;其中䗛蜢总科的碱基组成偏斜最强(AT skew = 0.23, GC skew = -0.23),而蜢总科碱基组成偏斜最弱 (AT skew = 0.11, GC skew = -0.14)。螽亚目 7 个总科的碱基偏斜均符合普遍规律,且 AT 偏斜相对弱于蝗亚目,AT 偏斜范围为 0.02~0.09 ;其中原螽总科的碱基组成偏斜最强烈 (AT skew = 0.09, GC skew = -0.29),蟋蟀总科 (AT skew = 0.08, GC skew = -0.28) 次之,而裂跗螽总科 (AT skew = 0.02, GC skew = -0.24) 最弱。
分别统计 167 种已测直翅目昆虫线粒体基因组蛋白质编码基因的密码子使用情况,计算 2 个亚目的相对同义密码子的使用频率 (relative synonymous codon usage, RSCU),统计结果如图 4 所示 :所有密码子中,UUA(L) 在蝗亚目和螽亚目中的 RSCU 值均为最高,分别为 2.91 和 2.74 ;GCG(A) 在蝗亚目和螽亚目中的 RSCU 值均为最低,分别为 0.11 和 0.22 ;UGG(W) 和 AUG(M) 在蝗亚目和螽亚目中均无偏好性 (RSCU = 1) ;密码子使用偏向性与密码子第三位点的 AT 偏向性呈现出一定相关性,第三位点为 A 或 U 的密码子使用频率普遍较高 ;同一个密码子的 RSCU 值在直翅目 2 个亚目昆虫间无显著差异。
2.4 直翅目昆虫线粒体蛋白质编码基因及进化速率
直翅目昆虫的 13 个线粒体蛋白质编码基因的 A+T 含量总体上略低于整个线粒体基因组(附表2)。
除脊蜢科,蛋白质编码基因的第三个位点 A+T 含量远高于密码子第一、二位点,例如测序物种数最多的剑角蝗科密码子的第三位点 A+T 含量高达 76.38%,而密码子第一位点和第二位点仅为 73.85% 和 72.13%。
通过比较,直翅目昆虫起始密码子具有以下特点 ( 附图 1) :atp6、cox2、cytB、nd3 和 nd4 的起始密码均为标准的三联密码子 ATN (ATA、ATT、ATC、 ATG) ;cox3、atp8、nd1、nd2、nd4、nd5 和 nd6 的起始密码子除了标准的三联密码子 ATN 以外,还有较特殊的起始密码子如 GTG、TTG、AGT、TTA、 CTG 和 CCT ;编码 cox1 基因的起始密码子最为复杂,除标准的三联密码子 ATN 作为起始密码子,大部分 cox1 使用了非标准密码子,例如 CCG、CAA、 ATT、TTA、ACC、ACG 等,此外不规则的四联密码子 ATGA[27]、GTGA[13]、ATAA[12] 等都是可能的 cox1 的起始密码子。这些特殊的起始密码子可以减小基因间隔区,同时又避免相邻基因发生重叠,同时可在转录成为 mRNA 后经过 RNA 编辑转换成正常的起始密码子并完成正常的翻译过程 [22, 28]。
与起始密码子不同,直翅目昆虫在终止密码子方面表现出较高的一致性 ( 附图 2) :atp8 的终止密码子为典型的三联密码子 TAA、TAG ;其余 12 种蛋白质编码基因的终止密码子除完整的三联体密码子 TAA 和 TAG 之外,还包括不完整的终止密码子如 TA 和 T。这种不完整的终止密码子在后生动物的线粒体基因组中很常见,研究者推测其可在转录后通过多聚腺苷酸作用形成完整的终止密码子进而完成转录终止 [29]。
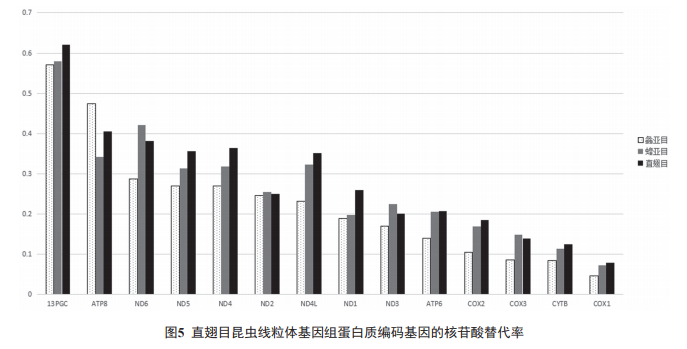
线粒体基因组外无组蛋白保护,容易受到代谢中间产物的诱变,相对于核基因组具有较高的进化速率 [30]。对直翅目 2 个亚目线粒体基因组的蛋白质编码基因进化速率 ( 用非同义替代率与同义替代率的比率 Ka/Ks 来表示进化速率 ) 进行对比,分析结果如图 5 所示 :不同的蛋白质编码基因的进化速率不同,其中 atp8 基因进化速率最快,而 cox1 最为保守,13 种蛋白质编码基因在直翅目中的进化速率顺序为 atp8 > nd6 > nd4 > nd5 > nd4L > nd1 > nd2 > atp6 > nd3 > cox2 > cox3 > cytB > cox1 ;相同的蛋白质编码基因在不同的类群中同样存在显著的差异,除 atp8 基因,其余 12 个蛋白质编码基因在蝗亚目中的进化速率均快于螽亚目 ;13 个蛋白质编码基因的 Ka/Ks 均小于 1,表现出纯化 ( 负 ) 选择,即基因发生非同义突变后作为劣势被淘汰了。
2.5 tRNA和rRNA特点
已测的直翅目昆虫线粒体基因组中,大多 trnSAGN 缺少 DHU 臂,缺失 DHU 臂后的 trnSAGN 仍可形成倒 L 型三级结构来维持 CCA 接受臂与反密码子间的距离 [31] ;其余 21 个 tRNA 均能折叠形成典型的三叶草结构,当然也有例外,霍山蹦蝗 (Sinopodisma houshana) 的 trnP 只有 58 bp,可变环和 TΨC 臂均不完整 [32]。tRNA 在形成三叶草结构碱基配对时会发生一些错配,主要是 G-U 错配。这种现象在昆虫中均普遍存在,在对蜘蛛 (Araneida) 线粒体 tRNA 的研究中发现,发生错配的碱基可以通过转录后编辑恢复正常配对,从而推测线粒体 tRNA 基因缺失 DHU 臂或 T 臂对其正常功能的行使影响不大 [33]。此外,直翅目昆虫 tRNA 携带的反密码子非常固定,尚未发现其他特殊的反密码子。
相关知识推荐:线粒体实验论文发表
rRNA 基因包括 rrnL 基因 (lrRNA 或 16S rRNA)和 rrnS 基因 (srRNA 或 12S rRNA),其大小和位置相对保守,且碱基组成表现出明显的 AT 偏好。 rrnL 基因的二级结构包括 6 个结构域 (I、II、III、 IV、V 和 VI),其中结构域 III 在节肢动物线粒体基因组中是缺失的,结构域 IV 和 V 保守程度较高 ; rrnS 基因的二级结构包括四个结构域 (I、II、III 和 IV),通常结构域 I 和 II 变异较大,而 III 和 IV 比较保守 [34]。直翅目昆虫线粒体基因组中 rrnL 基因位于 trnLCUN 和 trnV 之间,平均长度为 1 317 bp,其中最长的为短星翅蝗 (Calliptamus abbreviatus),长度为 1 555 bp ;最短的为欧洲巨蝼蛄 (Gryllotalpa pluvialis),长度为 1 236 bp[19]。直翅目昆虫线粒体基因组 rrnS 基因的长度远小于 rrnL 基因,平均长度仅为 800 bp,其中最长的为蛉蟋科的 Trigonidium sjostedti,长度为 1 004 bp[35],而最短的为暗色佛蝗 (Phlaeoba tenebrosa),长度仅为 212 bp。
2.6 控制区特点
直翅目昆虫控制区位置相对保守,通常位于 rrnS 和 trnI 之间,但也有例外,例如傅氏绿露螽、长裂华绿露螽和四川华绿露螽的控制区位于 nd2 和 trnQ 之间,翅糙颈露螽的控制区位于 trnY 和 cox1 之间 ;长度差异很大,其中最短的为疑钩顶螽 [14],仅有 70 bp,而最长的为长裂华绿露螽 [16],控制区长度为 3 122 bp。不同物种间控制区的长度变化主要与高度重复的序列的数量和大小相关,这些重复序列可进一步形成简单的发夹结构或更为复杂的二级结构,阻碍复制的进程。有研究表明,尽管不同物种线粒体 N 链的复制起点位置不同,在一些全变态昆虫的线粒体中存在的 T-strech (ploy-T > 10 bp) 可能与复制起始识别有关,而部分半变态昆虫的线粒体基因组并不含 T-strech 结构 [36]。在直翅目中,大部分蝗亚目以及螽亚目的蟋蟀总科和蝼蛄总科昆虫的线粒体控制区中虽然没有 T-strech 结构,但研究者在其对应位置发现 ploy-T 的结构被碱基 C 打断,并形成了相应的茎环结构,例如非洲飞蝗(Locusta migratoria) [12]、秦岭蹦蝗 (Sinopodisma tsinlingensis) [37]、僧帽佛蝗 (Phlaeoba infumata) [37]、小凸额蝗 (Traulia minuta) [37] 等 ;在螽亚目的螽斯总科几乎很难预测到这样的茎环结构 [34],但研究者在螽斯总科部分物种的线粒体控制区 N 链上发现了 T-strech 结构,例如摩门螽斯 (Anabrus simplex) [38]、笨棘颈螽 (Deracantha onos) [39]、优雅蝈螽 (Gampsocleis gratiosa) [38] 和黑角绿露螽 (Phaneroptera nigroantennata) [37]。总而言之,昆虫线粒体控制区的序列长度和结构变异很大。——论文作者:赵 乐1,2,李雪娟1 ,黄 原1 *
婢圭増妲�:閳剁姵鏋冮悮顔芥降閼奉亞鐓$純鎴欌偓浣烘樊閺咁喓鈧椒绔鹃弬鍦搼濡偓缁便垺鏆熼幑顔肩氨閿涘矁顕╅弰搴㈡拱閺傚洨灏炲鑼病閸欐垼銆冪憴浣稿灁閿涘本浼冮崰婊€缍旈懓锟�.閳垛€愁洤閺嬫粍鍋嶉弰顖欑稊閼板懍绗栨稉宥嗗厒閺堫剙閽╅崣鏉跨潔缁€鐑樻瀮閻氼喕淇婇幁锟�,閸欘垵浠堢化锟�鐎涳附婀虫い楣冩6娴滃牅浜掗崚鐘绘珟.

SCISSCIAHCI